コンピュータを駆使して新薬探索をスピードアップ

現代社会における主な課題の一つは、病気を治療する医薬品を開発する際の効率とコストです。新薬の研究開発は驚異的に進歩してはいますが、多くの製薬会社にとって、希少疾患分野に踏み出す場合の研究開発費は依然として高くなっています。「見捨てられた」疾患は多数存在しており、各疾患の患者は世界に少数しかいないものの、それでもかなりの数です。北野宏明教授率いるオープンバイオロジーユニットのクンイー・シーン研究員は、まさにこの問題に取り組んでいます。このたびオンラインジャーナル プロス・ワン(PLOS ONE)で発表された論文の中で、同研究員は薬剤となる可能性のある物質を特定するための計算論的アプローチについて説明しています。この技術は、新薬発見のスピードアップをはかりながらもその開発費用削減をもたらす可能性を持っています。
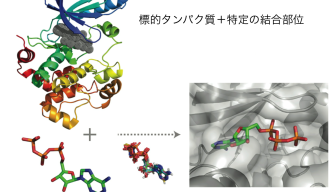
薬剤の有効成分である小分子とある種のタンパク質が相互作用するかを見極めるために、大学や製薬会社の研究者たちはいくつかの方法を用います。多くの疾患は、あるタンパク質の活性が強くなりすぎるか弱まることによって生じます。新薬発見の最終目標は、正常な働きをしなくなったタンパクを元に戻すと考えられる、小分子を発見することです。この過程は従来、実験室の中で、疾患の原因であることが知られているタンパク質に対し小分子一つ一つを手作業で検査することで実施されてきました。この方法ではコストも時間もかかります。コンピュータが登場すると、科学者たちは候補となる小分子を仮想的に特定してから、それらに対する実験室での検査に進めるようになりました。この方法の問題点の一つは、計算では情報の一片、すなわち小分子が特定のタンパク質に結合するかどうかという情報しか得られないことです。小分子が相互作用する可能性のある他のタンパクについてはわからないのです。そのような予期しない相互作用が、望ましくない結果、すなわち副作用をもたらす場合は問題となります。予期していなかった副作用は、患者が薬剤を使用する際のリスク増大や患者が負担する費用の増加という結果をもたらし、極端な場合には医薬品市場からの撤退につながります。
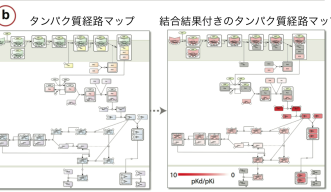
シーン研究員の研究が役に立つのはこの点です。同研究員はドッキング・シミュレーションと呼ばれる技術を使います。これはある小分子があるタンパクに結合するかどうかを、計算機の中で仮想的に実験し見極める技術です。ただし、通常行われているように、疾患の原因となっている一つのタンパクに対する結合性を多くの小分子を対象としてスクリーニングするのではなく、多くのタンパクに対する一つの小分子の結合性をスクリーニングするのです。その結果、オンターゲットおよびオフターゲットと呼ばれる、変化させたいタンパク質と、変化させたくないけれども小分子が予期せず相互作用してしまうタンパク質を発見することができます。これにより、実験段階に到達する前であっても、特定の薬剤の毒性を予測することができます。シーン研究員らは、複数のドッキング・シミュレーション・アルゴリズムを機械学習システムを併用して組み合わせることで、既存の方法と比べて、オンターゲットおよびオフターゲット候補をより効率よく予測できることを示しました。
シーン研究員の最終目標は、これらの計算論的手法を駆使して安価な新薬を創出し、より安全で患者が購入しやすい医薬品開発につなげることです。同研究員は、「現在この方法をより大きなシステムに適用する前に、小規模で予測結果を改善しているところです」と述べています。今後は東京大学医科学研究所と協力して、A型インフルエンザ・ライフサイクル・パスウェイマップ(FluMap)でシーン研究員のシステムを検証する予定です。同研究員は、OISTの高性能計算機(HPC)システム(TOMBO)の計算能力を用いて、実際の細胞環境により近い環境を仮想的に考慮することで、より進歩した予測技術を開発する予定です。
PLOS ONE誌掲載のシーン研究員の論文は下記リンク先から読むことができます。
http://www.plosone.org/article/info%3Adoi%2F10.1371%2Fjournal.pone.0083922
(エステス キャスリーン)
研究ユニット
広報・取材に関するお問い合わせ
報道関係者専用問い合わせフォーム